Das ExploreCOVID Projekt, welches gefördert wird vom Bundesministerium für Bildung und Forschung (BMBF), hat als Ziel die Analyse von Patientenanamnese sowie von klinischen und bildgebenden Daten über mehrere medizinische Zentren hinweg, um standardisierbare diagnostische Kriterien zu identifizieren. Dadurch wird eine umfassende Datenbank voller strukturierter Daten etabliert, die für zukünftige epidemiologische Erkenntnisse und Risikostratifizierung von COVID-19 Patienten genutzt werden kann.
Aktuelle Erkenntnisse deuten darauf hin, dass die Thorax-CT-Bildgebung ein wertvolles Hilfsmittel bei der Diagnose, Epidemiologie und Bewertung des Therapieansprechens von COVID-19-Patienten sein kann. Sie bietet eine hohe Sensitivität, kurze Durchlaufzeiten und eine breite Verfügbarkeit. Daher kann ein Thorax-CT RT-PCR-Tests ergänzen, insbesondere in Situationen mit unklarem klinischen Bild, wie z.B. bei einem negativen RT-PCR trotz starker anamnestischer Evidenz für COVID-19. Noch wichtiger ist, dass es die Möglichkeit bietet, den Krankheitsverlauf direkt zu beurteilen und eine Methode für die Beurteilung des Therapieansprechens bei anstehenden Studien mit neuen Therapeutika sein kann. Um es zu einem geeigneten Instrument zu entwickeln, ist jedoch ein standardisierter quantitativer Ansatz erforderlich. Darüber hinaus ist eine multizentrische Zusammenarbeit unerlässlich, um eine ausreichend große Datenbank mit einer gleichbleibend hohen Datenqualität zu erzeugen, die für moderne Data-Mining-Methoden von entscheidender Bedeutung ist.
Alle Zentren, die an diesem Projekt beteiligt sind, erhalten Zugriff auf die COVID-19-Funktionalität der mint Lesion™ Plattform. Die Studienzentren können bereits verfügbare Thorax-CT-Bilder strukturiert befunden, eine breite Palette bildbasierter Parameter (einschließlich Radiomics Parameter) extrahieren, sie mit anderen klinischen und anamnestischen Daten verknüpfen und dann die resultierenden strukturierten Daten an die Projektpartner übertragen, um die für die Diagnose und das Staging von COVID-19 relevanten Parameter zu ermitteln. Trotz der sehr hohen Anzahl unterschiedlicher Datenquellen gewährleistet dieser Ansatz, dass bei der Extraktion der Daten einheitliche und homogene Kriterien angewendet werden.
Die Schlussfolgerungen können sofort in die Befundungsvorlage von mint Lesion™ umgesetzt und weltweit zur Verwendung in Diagnoseverfahren und zur Beurteilung des Therapieansprechens in klinischen Studien zur Verfügung gestellt werden.
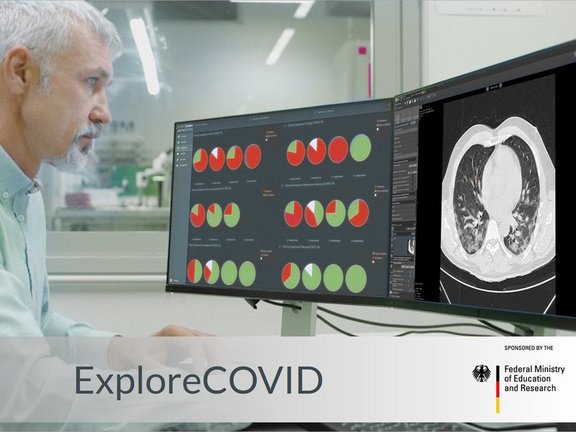
ExploreCOVID: Eine explorative Kohortenstudie zur Identifizierung optimaler CT-Bildgebungs-Biomarker in Kombination mit klinischen Markern und PCR-RT für die Diagnose und Beurteilung des Therapieansprechens von COVID-19
Ähnliche Inhalte
Ähnliche Inhalte
Setzen Sie auf KI-gestützte Lungenrundherden-Erkennung und -Analyse für eine umfassende Patientenversorgung
Entdecken Sie die Möglichkeiten eines KI-gestützten und optimierten Lungen-Screenings mit contextflow ADVANCE Chest CT integriert in mint Lesion.…
Vergleich von iRECIST und RECIST 1.1 zur Beurteilung der Immuntherapie bei Melanom und nicht-kleinzelligem Lungenkrebs
In einer retrospektiven Studie am Universitätsklinikum Köln wurden die radiologischen Kriterien iRECIST und RECIST 1.1 zur Beurteilung des…

Universitätsklinikum Köln: Vergleich von iRECIST und RECIST 1.1 zur Beurteilung der Immuntherapie bei Melanom und nicht-kleinzelligem Lungenkarzinom
In der am Universitätsklinikum Köln durchgeführten retrospektiven Studie wurden zwei Kriterien zur radiologischen Beurteilung des…